Integrative Virologie
Leitung: Prof. Dr. Maya Topf
Die Anheftung von Viren an die Zelle, ihr Zelleintritt, ihr Entpacken (Uncoating), die Biosynthese, Assemblierung und die Freisetzung - alle diese Schritte im Infektionszyklus erfordern die Bildung von Protein-Protein-Interaktionen zwischen den viralen Proteinen selbst sowie zwischen viralen und Wirtsproteinen. Daher ist es von großem biomedizinischen Interesse, die Struktur der Komplexe zu verstehen, die durch diese Interaktionen gebildet werden. Ziel ist es dabei, antivirale Therapien zu entwickeln, welche die Bildung dieser Komplexe blockieren können, oder alternativ Viren für den Einsatz als zielgerichtete Trägersysteme umzugestalten. In den letzen Jahren hat die Verwendung von integrativen, informationsgetriebenen Ansätzen zur Modellierung von Strukturen von Komplexen, wie sie z.B. von Viren in Zellen gebildet werden, an Popularität gewonnen. Die Abteilung Integrative Virologie wendet einen integrierten systembiologischen Ansatz an, der rechnergestützte und experimentelle Methoden kombiniert, die speziell darauf ausgerichtet sind, ein umfassendes Bild von Virusinfektionen zu erhalten; mit besonderem Schwerpunkt auf KryoEM, massenspektrometriebasierter Proteomik sowie Bioinformatik.
Arbeitsgebiete
Validierung und Verfeinerung des 3D-EM-Modells
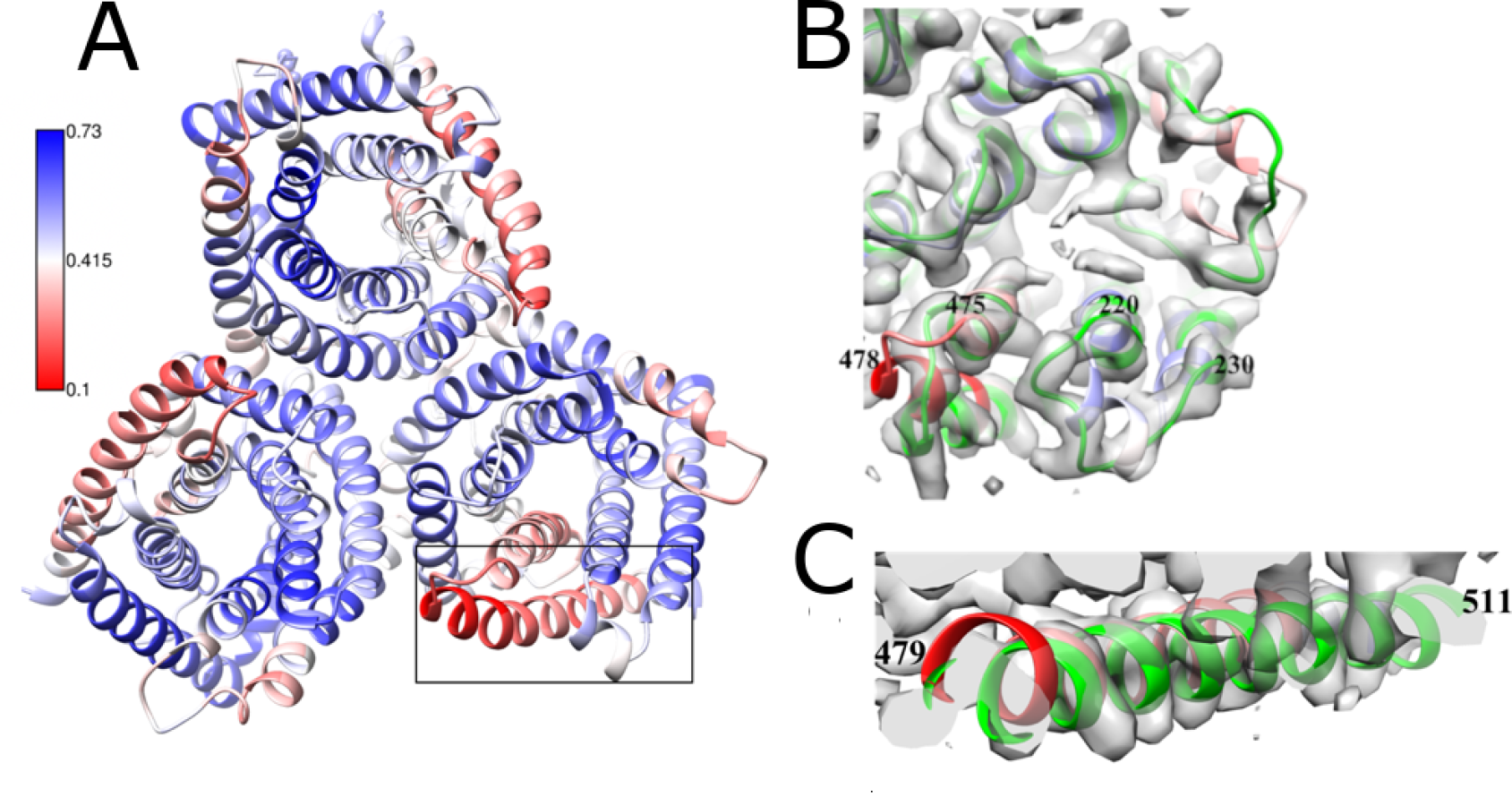
Die Abteilung entwickelt Tools zur Anpassung und Bewertung von Atommodellen in Kryo-EM-Karten. Diese Methoden werden zur Charakterisierung der makromolekularen Assemblierung von Viren eingesetzt. Die Abteilung hat die Anpassungs- und Validierungssoftware TEMPy entwickelt. Mit Hilfe von Kryo-EM hat die Gruppe kürzlich die Protein-Protein-Interfaces in makromolekularen Assemblierungen untersucht. Mithilfe einer auf Machine Learning basierenden Metrik (PI-Score) wurden 5873 Interfaces in 1053 PDB hinterlegten atomistischen Kryo-EM-Modellen (einschließlich SARS-CoV-2-Komplexen) sowie in Modellen, die für die CASP13-Kryo-EM-Ziele und den EM-Modellwettbewerb eingereicht wurden, bewertet.
Joseph et al. Methods 2016, Cragnolini Acta D 2021, Malhotra et al. Nat. Commun. 2021
Massenspektrometrie-basierte Modellierung
Die chemische Cross-Linking-Massenspektrometrie (XL-MS) ist eine Technik, die lösungsmittelzugängliche Abstandshalter für Proteine und Proteinkomplexe in Lösung zur Verfügung stellt. Das Verfahren erzeugt vier verschiedene Arten von Peptiden: Cross-Links, Mono-Links, Loop-Links und unmodifizierte Peptide. Die Abteilung hat eine Bewertungsfunktion mit der Bezeichnung Matched and Non-accessible Cross-Link (MNXL) entwickelt, um Modelle der Proteinstruktur auf der Grundlage von Cross-Linking-Informationen zu bewerten, wobei sowohl die Lösungsmittelzugänglichkeit der verknüpften Reste als auch der Abstand zwischen den beiden verwendet wird. Mono-Links liefern nur Informationen über die Lösungsmittelzugänglichkeit, sind aber in einem XL-MS-Experiment viel häufiger anzutreffen als Cross-Links. Daher wurde zudem ein Ansatz zur Bewertung von Proteinmodellen anhand von Mono-Links entwickelt. Der kombinierte Cross-Link-Mono-Link-Score heißt XLMO.
Matthew Allan Bullock et al. MCP 2016; Bullock et al. 2018 Structure, Bioinformatics; Sinnott et al. Structure 2020
Protein-Protein-Interaktionsnetzwerke
Die Abteilung nutzt Protein-Protein-Interaktions (PPI)-Netzwerkanalysen, um Virusfamilien neu funktional zu charakterisieren und hat ein computerbasiertes Framework für den Aufbau von PPI-Netzwerken entwickelt, das sowohl experimentell validierte als auch rechnerisch vorhergesagte PPI-Daten kombiniert. Mithilfe dieses Frameworks wurden auf Systemebene Kompilationen der binären Interaktionen zwischen viruskodierten Proteinen erstellt. Dies ist auf drei verschiedene Arten von menschlichen Herpesviren angewandt worden. Mithilfe des Consensus-Clustering-Ansatzes wurden die Community-Struktur der Netzwerkdaten untersucht und es konnten funktionelle Assoziationen höherer Ordnung zwischen viralen Proteinen gezeigt werden. In Komination mit der primären Sequenzanalyse einzelner Proteine brachte diese Studie neue funktionelle Erkenntnisse über zuvor unzureichend charakterisierte Proteine.
Hernández Durán et al, PLoS Biol 2019, Hernández Durán et al mSystems 2019