Proteinstrukturen besser verstehen
Donnerstag, 17.12.2020
Ein Interview mit CASP14 Organisatorin Maya Topf
Die neueste HPI/CSSB-Abteilungsleiterin, Prof. Maya Topf, ist eine von insgesamt fünf Organisatorinnen und Organisatoren des „14. Community Wide Experiment on the Critical Assessment of Techniques for Protein Structure Prediction” (CASP14), das virtuell vom 30. November bis zum 4. Dezember 2020 stattgefunden hat. Zu Beginn der Konferenz am 30. November wurde bekannt gegeben, dass das vom Londoner KI-Labor DeepMind entwickelte Programm AlphaFold dazu in der Lage ist, die Form vieler Proteine mit einer vergleichbaren Genauigkeit wie im Labor zu bestimmen.
In einem kurzen Interview berichtet Maya Topf von der Erfahrung, Teil der CASP14 zu sein und über die Auswirkungen, die AlphaFold auf die wissenschaftliche Gemeinschaft haben wird:
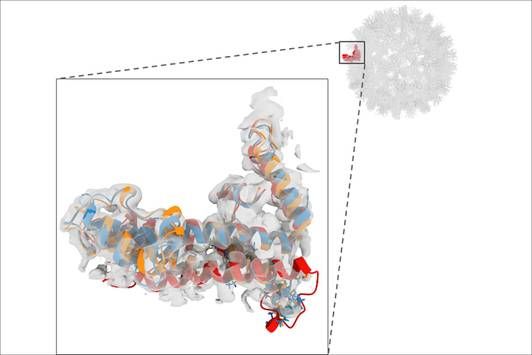
Was ist das CASP-Experiment?
CASP ist ein echtes Gemeinschaftsprojekt, an dem über 100 Gruppen aus der ganzen Welt teilnehmen. Die Gruppen erhalten die Aminosäuresequenzen von etwa 100 Proteinen und werden gebeten, die Struktur des Proteins mithilfe von Computersimulationen vorherzusagen. Diese Strukturen wurden bereits experimentell bestimmt, sind aber noch nicht veröffentlicht worden.
Wie lange sind Sie schon dabei?
Dies ist das zweite Mal, dass ich das CASP-Experiment organisiere. Es gibt eine zunehmende Anzahl von Kryo-EM- Zielstrukturen, die jetzt Teil der Herausforderung sind. Da dies das Fachgebiet meiner Abteilung ist, wurde ich 2017 angesprochen, dem Organisationskomitee beizutreten. In diesem Jahr wurden 7 % der Zielstrukturen experimentell mit Einzelteilchen-Kryo-EM bestimmt.
Haben die diesjährigen Organisatoren die Ergebnisse von AlphaFold erwartet?
AlphaFold hat auch CASP13 "gewonnen". Das Programm war sehr gut, also haben wir erwartet, dass es auch dieses Jahr gut abschneiden würde. Aber wie gut es schließlich abgeschnitten hat, war eine komplette Überraschung.
Welche Auswirkungen wird AlphaFold auf die Forschungsgemeinschaft haben?
Die Auswirkungen werden enorm sein, wenn wir verstehen und wiederholen können, was DeepMind getan hat. Wir wären in der Lage, die Struktur von einkettigen Proteinen auf einem Level vorherzusagen, das nahe an das experimentelle heranreicht. Aber es ist noch nicht klar, ob DeepMind seine Methoden offenlegen wird.
Hoffentlich werden andere Gruppen die Methodik von AlphaFold verstehen und ähnliche Programme entwickeln, die dann der wissenschaftlichen Gemeinschaft zur Verfügung gestellt werden. Das ist so nach CASP13 geschehen. Die Top-Gruppen auf dem Gebiet waren dazu in der Lage, die DeepMind-Methode zu lernen und sogar zu verbessern. Dieses Mal haben die internationalen Top- besser abgeschnitten als AlphaFold bei CASP13.
Wie wird AlphaFold die Infektionsforschung beeinflussen?
Wenn wir die Struktur eines Proteins besser verstehen, haben wir auch ein besseres Verständnis über dessen Funktion. Dies könnte zu einem verbesserten Wirkstoffdesign und neuen Angriffspunkten für Medikamente führen.
Das hat Auswirkungen auf den gesamten Bereich der Strukturbiologie. Es ist erstaunlich - jetzt können wir jede Sequenz nehmen und die Proteinstruktur bestimmen. Das wird unser Verständnis allgemeiner Krankheitsmechanismen verbessern.
Ist dies das Ende der experimentellen Strukturbiologie?
Nein, auch wenn AlphaFold einzelne Proteinstrukturen vorhersagen kann, kann es nicht vorhersagen, wie die Proteine in großen Komplexen interagieren. Proteine funktionieren nicht isoliert und wir sind noch weit davon entfernt zu verstehen, wie sie in Zellen funktionieren. AlphaFold ist ein Schritt nach vorne - die Methode ist gut darin, einzelne Proteinstrukturen vorherzusagen, aber nicht ihre Dynamik und ihre Interaktionen mit anderen Proteinen. Es werden auch weiterhin experimentelle Daten benötigt.
Wie wird AlphaFold Ihre eigene Forschung beeinflussen?
Ein Teil der Forschung meiner Gruppe umfasst die Vorhersage der Strukturen großer Proteinkomplexe. Um dies zu erreichen, müssen wir zunächst die Struktur der einzelnen Komponenten modellieren. Wenn die Modelle der Komponenten genauer sind, dann werden die Modelle des gesamten Komplexes letztendlich auch genauer sein.
Wie haben Sie sich dabei gefühlt, ein Teil von all dem zu sein?
Es war wirklich aufregend! Ich habe gespannt auf die Nachricht gewartet und wusste, dass sie einen großen Sturm in der wissenschaftlichen Gemeinschaft auslösen würde. Das ist eine große Sache - das Problem der Proteinstrukturvorhersage ist im Wesentlichen gelöst. Wissenschaftlerinnen und Wissenschaftler haben viele, viele Jahre daran gearbeitet. Wir dachten eigentlich, dass es noch viel länger dauern würde, diesen Grad an Genauigkeit zu erreichen. Unterm Strich wird es also die wissenschaftlichen Gemeinschaft weiter voranbringen.
Insgesamt war es eine tolle Erfahrung, dabei zu sein, und das nicht nur wegen der Aufregung um AlphaFold. Es war toll, die Kategorien für den Wettbewerb vorzubereiten, die Ziele festzulegen und mit den Gutachtern zu arbeiten. Meine Gruppe war auch Gutachter bei den letzten beiden CASP-Experimenten. Wir mussten auswerten, wie gut die Teilnehmenden bei den Kryo-EM-Targets abgeschnitten haben.
Wie wird CASP15 aussehen?
Wir hoffen, dass wir mehr Targets von Proteinkomplexen haben werden, da dies ein Problem ist, das noch nicht gelöst wurde. Dafür brauchen wir mehr Kryo-EM-Wissenschaftler, die uns Targets liefern. Potenziell wird es Interesse an der Vorhersage der Proteindynamik geben - wir werden noch größere Probleme angehen.